2025年8月29日,《自然·通讯》(Nature Communications)正式发表了由武汉大学化学与分子科学学院周翔教授/翁小成教授团队、刘艺斌教授课题组、武汉大学医学研究院胡致远教授课题组、武汉大学中南医院消化内科王红玲主任团队共同完成的一项研究成果。论文题目为“Whole-genome methylation profiling of extracellular vesicle DNA in gastric cancer identifies intercellular communication features”。武汉大学周翔教授/翁小成教授团队的博士后林冰倩博士、胡致远教授课题组的焦振娜与武汉大学中南医院消化内科王红玲主任团队的董守权医生为论文共同第一作者,周翔教授、刘艺斌教授、胡致远教授、王红玲教授和翁小成教授为论文的共同通讯作者,武汉大学为唯一通讯单位。

外泌体(EVs)作为癌症诊断和预后研究中的潜力生物标志物,正引起广泛关注。然而,基于EV-DNA甲基化的液体活检技术在临床应用中仍处于探索阶段。由于EV-DNA的含量较低且片段较长,在临床样本中进行全基因组甲基化分析具有一定挑战性。针对这一问题,研究团队开发了一种创新的Tn5辅助酶促甲基化测序技术——TEMPT,该技术结合了单接头Tn5转座酶与转化后加尾技术,并联合酶促转化法(Em-seq)。通过这一方法,成功实现了微量EV-DNA样本的全基因组甲基化测序,同时避免了传统亚硫酸盐处理带来的各种问题。
研究团队对58例临床胃癌和胃息肉样本进行了EV-DNA全基因组甲基化测序,结果展示了TEMPT方法卓越的重复性。在发现队列和验证队列中,该方法均能稳定、可靠地区分癌症样本与胃息肉样本。通过分析差异甲基化区域(DMRs),研究还识别了多个潜在的肿瘤相关生物标志物,进一步强调了外泌体在细胞间通讯中的关键作用。此外,研究人员还发现EV-DNA的特征与免疫细胞相关,提示免疫细胞在EV-DNA释放过程中也发挥了重要作用。通过细胞去卷积分析,研究团队首次揭示了EV-DNA的组织来源(TOO)及其免疫细胞组成。
这项研究通过TEMPT技术对EV-DNA进行全基因组深度测序,显著丰富了现有的cfDNA和EV-DNA突变研究。随着样本队列的扩大,未来将进一步完善EV-DNA甲基化参考数据库,并提升计算模型的准确性,从而实现更加精准的分析。同时,解决EV异质性问题将有助于提高EV-DNA分析的准确性。未来,结合TEMPT方法与CUT&TAG技术,不仅有望深入分析EV-DNA的甲基化特征,还能够探索其上的组蛋白修饰。这些进展将进一步增强TEMPT技术在临床应用中的价值,并拓展其潜在的应用领域。
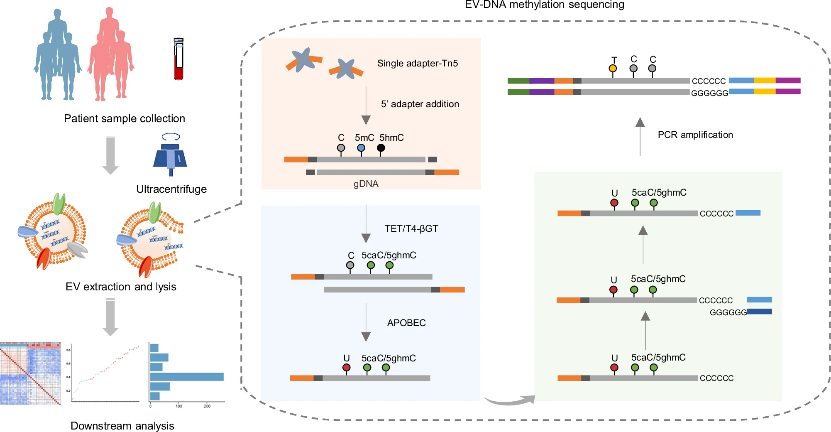
图1. 用于EV-DNA样本深度甲基化测序的TEMPT概述
论文链接:https://www.nature.com/articles/s41467-025-63435-w